Como nova informação genética é gerada durante o processo evolutivo?
- Atualizado no dia 17 de abril de 2023 -
Compartilhe o artigo:
Um argumento anti-evolucionista bastante comum baseia-se na suposta impossibilidade de novos genes emergirem e serem acumulados, ou seja, que o material genético dos seres vivos supostamente só poderia ser reduzido ou mantido. Como explicar, por exemplo, que a espécie de planta Glycine max carrega mais de 50 mil genes codificadores de proteínas enquanto uma bactéria do gênero Candidatus chega a ter menos de 200 genes codificadores? Se ambas apresentam um ancestral em comum, isso significa que houve um gigantesco acréscimo de informação genética no DNA da planta. Segundo os anti-evolucionistas, mutações no máximo reduzem informação genética, sendo impossível aumentá-la via processos naturais. Ora, nesse caso, seria necessário algo sobrenatural construindo e adicionando os genes para o acúmulo de novidades genotípicas e fenotípicas, certo?
A resposta seria 'sim' para esse "problema", caso o indivíduo possua um conhecimento muito limitado de genética e não acompanhasse os periódicos científicos - como parece ser a realidade daqueles que rejeitam a evolução biológica. Aliás, recentemente os pesquisadores conseguiram decifrar o complexo genoma do trigo (T. aestivum) (1), o qual mostrou ser constituído de 3 sub-genomas combinados de outras espécies e conter 85% de elementos repetidos. Opa, hibridização somando genomas e duplicações genéticas! Quando se analisa o material genético dos seres vivos, é impossível descartar o contexto evolutivo.
- (1) Para mais informações, acesse: Cientistas finalmente conseguem decifrar o genoma do trigo
IMPORTANTE: Para a melhor compreensão do artigo a seguir, fica a recomendação de leitura (conceitos básicos de genética): Novas letras artificiais para o nosso maquinário genético
- Continua após o anúncio -
NOVOS GENES
As ramificações da árvore evolucionária deixam explícito uma vasta diversidade biológica, com a enorme variedade genômica a fonte essencial e primária para essa riqueza fenotípica. A evolução do genoma é dirigida por vários fatores, como mudanças incrementais nas sequências codificadoras e não-codificadoras, assim como na emergência de novos genes. O crucial papel dos novos genes como principais fomentadores da evolução biológica (2) e da diversidade fenotípica é claramente indicado pela grande variedade nos números e tipos de genes entre as espécies.
- (2) Leitura recomendada: A Evolução Biológica é um FATO CIENTÍFICO
Ao longo da evolução, genes estão constantemente sendo adicionados e deletados do genoma, levando a mudanças na frequência de genes dentro de uma população (o significado de processo evolutivo a nível populacional). Cada gene em um organismo 'nasceu' em determinados períodos na evolução: alguns são antigos, alguns são jovens e muitos estão ativamente no processo de serem gerados ou perdidos. Dentro de um grupo de espécies em próximo parentesco, 'novos genes' podem ser definidos como aqueles presentes em todos os membros de um grupo monofilético mas ausentes em todas as espécies fora desse grupo. A origem de novos genes trazendo novas informações genéticas é um processo microevolucionário. Primeiro, uma estrutura protogenética é gerada por uma mutação no genoma de uma única célula germinativa. Essa estrutura protogenética precisa então ser espalhada através de uma população até ser fixada. Nesse sentido, entram os mecanismos evolutivos, como seleção natural e deriva genética, para esse serviço de propagação.
Tanto antes quanto depois da fixação, o protogene acumula mutações que o conferem novas estruturas e funções benéficas, às vezes novas, as quais, então, se submetem à seleção natural. A partir do ponto em que o protogene carrega uma função otimizada e é fixado no genoma, ele será essencialmente o mesmo como a maioria dos outros genes mais antigos no genoma e pode ser considerado um novo gene.
- Continua após o anúncio -
PRODUÇÃO E ACUMULAÇÃO DE NOVOS GENES
Novos genes comumente surgem através da duplicação de genes pré-existentes, seja no nível do DNA ou pela retro-transposição através de um RNA intermediário. Nesses casos, o novo gene pode manter funções similares ao gene ancestral ao longo do período evolucionário ou pode passar por um processo de diversificação até uma completa nova função evolua. Novos genes com novas funcionalidades podem também se originar de novo a partir de DNA não-codificante ou pode ser derivado de várias alterações e rearranjos em loci genéticos pré-existentes. Além disso, novas expressões e funcionalidades genéticas requerem elementos regulatórios que podem pré-existir no novo locus do gene ou que podem precisar evoluir.
Mutação é o primeiro passo fundamental para a formação de novos genes, e hoje cerca de 12 mecanismos moleculares são bem conhecidos e descritos para a ocorrência desse processo. Apesar de muitos associarem mutações genéticas apenas com prejuízos ao organismo e deformidades, muitos mecanismos mutantes fornecem uma escada essencial para o processo evolucionário.
1. Duplicação genética Esse provavelmente é o mais prevalente mecanismo fomentando a formação de novos genes. Durante mutações cromossômicas, às vezes uma cópia extra de alguma região do DNA contendo um gene é duplicada. Essas duplicações podem surgir principalmente como produto de vários tipos de erros na replicação do DNA e no maquinário genético de reparação. As regiões duplicadas podem estar localizadas adjacentes umas às outras ou uma das regiões duplicadas pode estar em sua localização normal e a outra em uma nova localização em uma diferente parte do mesmo cromossomo ou mesmo em outro cromossomo. Fontes comuns de duplicações genéticas incluem crossing-over assimétrico, retro-transposição, poliploidismo e transposição do DNA duplicado.
- Crossing-over assimétrico
Esse processo produz sequências repetidas sucessivas e contínuas de DNA. Dependendo da posição de cossing-over (cruzamento), as regiões duplicadas podem conter parte de um gene, um gene inteiro, ou vários genes
- Retro-transposição
Esse é um processo de duplicação onde o RNA mensageiro (mRNA) de um gene é transcrito de forma reversa para um DNA complementar (cDNA) e, então, inserido no genoma. Existem várias características moleculares de retroposição: falta de íntrons e sequências regulatórias de genes, presença de sequências poli-A e presença de pequenas repetições diretas divisórias. A principal diferença desse mecanismo para o crossing-over assimétrico é a presença de íntrons. Íntrons são as pequenas sequências de DNA presentes entre as sequências codificadoras que se dividem após a transcrição. Se íntrons estão presentes nos genes originais, eles estão também presentes nos genes duplicados através de cruzamentos assimétricos, mas ausentes em retro-genes. Um gene duplicado gerado por retro-transposição é geralmente não-ligado ao gene original, já que a inserção de cDNA dentro do genoma é relativamente aleatória.
- Transposição duplicativa
Esse mecanismo pode ser alcançado por um de dois caminhos: recombinação não-alélica homóloga (NAHR) ou junção não-homóloga final (NHEJ). A diferença entre os dois caminhos é baseada no uso ou não de sequências homólogas como template (modelo) durante o reparo de quebra da dupla cadeia, e essa diferença pode também ser usada para inferir o mecanismo pelo qual genes individuais são duplicados. A recombinação entre essas sequências homólogas não-alélicas pode resultar na duplicação de sequências interventoras, algo que pode, então, levar a mais duplicações por causa do pareamento entre os novos parálogos (genes que compartilham uma mesma ancestralidade mediada por duplicações; são cópias que ganham novas funções e, por isso, acabam sendo mantidas no genoma).
- Poliploidização
Esse é um processo evolucionário onde dois ou mais genomas são unidos pelo mesmo núcleo - geralmente durante hibridizações entre espécies seguido de duplicações cromossômicas. Duplicações genômicas inteiras seguidas por uma massiva perda de genes e especialização têm se mostrado muito importantes para a ocorrência de grandes eventos evolucionários entre vários grupos de organismos, especialmente entre as plantas e os fungos. Entre as plantas, é estimado que o poliploidismo ocorreu em pelo menos 70% das linhagens das angiospermas (3) e em 95% das linhagens das pteridófitas.
- (3) Leitura recomendada: Genoma completo da abóbora é sequenciado e revela estranho processo evolucionário
O ponto mais importante das duplicações genéticas, especialmente no poliploidismo, é o fornecimento de amplo material genético extra onde os mecanismos evolutivos podem atuar com maior facilidade e com um maior espectro de possibilidades. Ora, se temos mais cópias do que o necessário de um gene ou novas cópias adicionadas de uma fonte genômica terceira, as cópias e genes excedentes podem passar por processos de mutações e seleções naturais sem prejudicar o organismo, já que as cópias normais cumprirão as funções básicas necessárias para a sobrevivência. Essas mutações nas cópias extras podem render novos genes associados a novas expressões fenotípicas, gerando um acúmulo de informações genéticas positivamente selecionadas e preservadas no genoma. Além disso, eventos de duplicação genômica inteira já geram um indivíduo descendente com um fenótipo substancialmente diferente, ou seja, um potencial passo inicial de especiação.
A vantagem e o fomento evolutivos conferidos pelo poliploidismo foram reforçados e demonstrados experimentalmente em um estudo de 2018 publicado na Nature (Ref.30) que analisou genomicamente a espécie Arabidospis kamchatica, uma planta tetraploide (alo-poliploide) altamente adaptativa a regiões diversas tanto no Hemisfério Sul quanto no Hemisfério Norte. Outro exemplo mais recente foi detalhado em um estudo publicado no periódico eLife (Ref.31), onde duplicações em gramíneas - levando a 14 cópias do gene FTL9 [FLOWERING LOCUS T (FT1) paralog FT-like9], associado com o florescimento nas angiospermas - permitiram a mutação de uma das cópias e a evolução de um importante novo traço adaptativo. Muitas plantas, como beterraba e cenoura, florescem no verão, com essa programação sendo ativada pelas baixas temperaturas do inverno em um processo chamado de vernalização, baseado na expressão do gene FTL9. No entanto, gramíneas como arroz e trigo, conseguem usar os dias mais curtos do inverno como gatilho de programação através de um alelo do FTL9 que possui a deleção de um único nucleotídio (no caso, uma guanina), alterando a estrutura e a funcionalidade da proteína FTL9 expressa. Isso permite com que em regiões de clima mais quente, onde as temperaturas no inverno não caem tanto, a programação para o verão seja feita de forma mais eficiente.
Vários genes já foram observados sendo duplicados naturalmente em laboratório e diversos outros em espécies diversas já foram apontados como oriundos de processo de duplicação, incluindo vários exemplos no corpo humano. Aliás, evidências se acumulando (Ref.7) reforçam a hipótese de que a origem dos vertebrados no planeta foi catalisada por um evento duplo de duplicação genômica inteira, dando origem a um ancestral octoploide, o qual, desde então, passou por sucessivas reduções genômicas ao longo do percurso evolutivo até o genoma dos vertebrados ser 'diploidizado' novamente. Durante esse processo, a emergência de diversas importantes inovações morfológicas e mecanismos regulatórios (DNA não-codificante e processos epigenéticos) foram favorecidos, ajudando a determinar a grande complexidade e variedade dos grupos vertebrados (Ref.17). Outros grandes "pulos"/avanços evolutivos também são explicados pelas duplicações genômicas. Constantemente em periódicos são publicados estudos descrevendo novos eventos de duplicações genéticas, especialmente após o sequenciamento genômico de plantas. Alguns estudos estimam que cerca de 80% dos novos genes surgem desse processo.
- Um interessante caso de triploide gerado via duplicação genômica, representando uma recente evolução: Super-espécie parece ter surgido acidentalmente em um aquário e está ameaçando a biodiversidade ao redor do mundo
- O grande sucesso evolucionário das angiospermas também está associado com duplicações genômicas: Resolvendo o abominável mistério evolucionário das angiospermas
Clássicos exemplos de duplicações genéticas conferindo novos fenótipos foram demostrados a partir da década de 1990 em moscas do gênero Drosophila. Para exemplificar, a mutação dominante Bar no cromossomo X da Drosophila produz um olho na forma de fenda ao invés de um normal e oval. O efeito é produzido pela redução do número de facetas oculares. Estudos citológicos dos cromossomos politenos mostraram que o fenótipo Bar é causado por um processo de duplicação crossing-over assimétrico da região cromossômica chamada de 16 A.
Gametas contendo a deleção geralmente morrem ou produzem zigotos inviáveis. Já gametas contendo a duplicação produzem filhotes Bar. Machos carregando a duplicação Bar em um estado hemizigoto expressam olhos severamente reduzidos. Fêmeas heterozigotas com um cromossomo Bar e um normal possuem olhos ligeiramente reduzidos.
Existe um outro tipo de erro/mutação genética que leva ao surgimento de duplicações que resultam em um número anormal de cromossomos: aneuploidismo. Nos mamíferos, esse processo é frequentemente prejudicial, levando a abortos espontâneos. Mas o aneuploidismo também pode gerar descendentes viáveis, como ocorre, por exemplo, na Síndrome de Down, onde uma cópia extra do cromossomo 21 é gerada. Aliás, nos humanos, eventos de duplicação em genes de regulação associados aos neurônios excitatórios e gliais radiais marcaram de forma importante nossa evolução desde a divergência com outras linhagens de primatas superiores (Ref.16).
EXEMPLOS NOTÁVEIS DE DUPLICAÇÕES GENÔMICAS
Com o grande avanço tecnológico na área de genética nas últimas décadas, cada vez mais genomas estão sendo sequenciados, revelando diversos eventos de duplicação genética que marcaram a história evolutiva de espécies diversas. De fato, você não explica enormes genomas ou diversos padrões genômicos sem o mecanismo evolutivo de duplicação genética e/ou sem hibridizações. Fora do contexto evolutivo, o material genético das angiospermas (plantas com flores), por exemplo, se torna ilógico.
TRIGO - Em um enorme esforço conjunto envolvendo 200 cientistas e 73 instituições de pesquisa em 20 países, foi realizado um sequenciamento de alta qualidade de 94% do genoma do trigo (Triticum aestivum L.), o qual engloba 21 cromossomos (Ref.26). Todo esse esforço justifica-se pelo fato de que o genoma hexaploide do trigo é altamente complexo e composto por 3 subgenomas - derivados da hibridização entre três espécies diploides -, com um tamanho mais de 5 vezes maior (16 Gb) do que o genoma humano, 107891 genes até o momento identificados e com mais de 85% de sequências repetidas (incluindo 3968974 cópias de TEs pertencentes a 505 famílias). Eventos de expansão genômica via duplicações foram observadas entre 8592 famílias de genes, sendo 6216 delas expandidas nos três subgenomas; apenas 78 famílias de genes sofreram reduções genômicas. Modificações epigenéticas também marcam substancialmente o genoma do trigo ao longos dos últimos 10 mil anos de domesticação (Ref.27).
O trigo fornece cerca de 20% do total de calorias e proteínas consumidos por humanos ao redor de todo o mundo, sendo também uma importante fonte de vitaminas e minerais. O cultivo dessa planta provavelmente determinou a transição do Neolítico para as sociedades sedentárias agrárias no Crescente Fértil. Para atender a demanda populacional em 2050, onde é projetado um total de 9,6 bilhões de pessoas, a produção de trigo precisará aumentar em 60%, e para alcançar isso preservando a biodiversidade, recursos hídricos e conteúdo nutricional, melhoramentos via ferramentas genéticas serão necessários.
CANA-DE-AÇÚCAR Fornecendo importante matéria-prima para a produção industrial de açúcar (sacarose) - 80% do total global - e de biocombustíveis*, a cana-de-açúcar (Saccharum spp.) é outro exemplo de genoma muito complexo, por ser altamente poliploide, aneuploide (diferentes números de cópias entre diferentes cromossomos), heterozigótica (origem bi-específica dos cromossomos) e possuir cromossomos recombinantes interespecíficos. O genoma desse gênero possui um tamanho em torno de 10 Gb e cerca de 25316 genes codificantes de proteínas, e é derivado a partir de sucessivas hibridizações entre duas espécies há centenas de anos (S. officinarum e S. spontaneum), ambas também altamente poliploides. Com 80 cromossomos no genoma (2n), a S. officinarum possui um alto conteúdo de açúcar e parece ter sido domesticada há cerca de 8 mil anos da espécie selvagem S. robustum. Já a S. spontaneum é uma espécie selvagem com vários citotipos e várias formas aneuploides (40 a 128 cromossomos), e que foi usada para a incorporação de resistência a doenças, vigor e adaptabilidade. Através de cruzamentos e de retro-cruzamentos entre essas duas espécies, foi obtido uma espécie altamente heterozigótica, aneuploide e com 100-130 cromossomos, a maioria derivados da S. officinarum, 10-20% da S. spontaneum, e ~10% de recombinantes interespecíficos (Ref.29).
----------
*Com mais de 7 milhões de hectares plantados e uma produção anual de 480 milhões de toneladas, o Brasil é o maior produtor mundial de cana-de-açúcar e líder em tecnologia de etanol.
-----------
QUIUÍ A fruta da espécie Actinidia chinensis - popularmente conhecida como Quiuí - possui um alto e singular conteúdo de vitamina C. Um estudo recente, publicado no periódico iScience (Ref.32) mostrou que dois eventos de duplicação - no caso, duas tetraploidizações - ocorreram nos ancestrais do A. chinensis dentro da família Actinidiaceae, introduzindo dois subgenomas em cada um deles. O primeiro evento de tetraploidização ocorreu há 50-57 milhões de anos e o outro há 18-20 milhões de anos. As cópias extras de genes geradas nesses eventos expandiram importantes genes funcionais, incluindo genes expressando instruções para a biossíntese e a reciclagem de vitamina C. Como a taxa evolucionária não mostrou-se muito acelerada - como normalmente ocorre com os eventos de duplicação via hibridização - fica sugerido que essas expansões refletiram uma auto-poliploidização, ou seja, eventos espontâneos de duplicação. Para essa última hipótese, os pesquisadores argumentaram que múltiplas cópias de cromossomos homólogos na auto-tetraploidização causa herança multissômica, a qual pode prolongar o tempo de ciclo celular, diminuindo a taxa de formação de novas gerações.
ÓPIO Publicado na Science (Ref.33), o sequenciamento do genoma do ópio (Papaver somnifeum L.) revelou 2,72 Gb distribuídas em 11 cromossomos, e uma assinatura de duplicação genômica inteira há cerca de 7,8 milhões de anos, e uma provável duplicação (ões) genética (s) há cerca de 100 milhões de anos durante a divergência das famílias Papaveraceae e Ranunculaceae. No estudo, os pesquisadores mostraram como duplicações, rearranjamentos e fusões de genes levaram à evolução de produtos metabólitos especializados na papoula do ópio, como a morfina.
 COALA Em 2018, o sequenciamento do genoma do Coala (Phascolarctos cinereus), um marsupial nativo da Austrália pertencente à família Phascolarctidae, foi publicado na Nature Genetics (Ref.35). Esses mamíferos se alimentam quase que exclusivamente da planta Eucalyptus spp., uma dieta que seria tóxica ou fatal para a maioria dos outros mamíferos. Com 16 cromossomos, o genoma dos coalas é um pouco maior do que o dos humanos, com 3,4 bilhões de pares de bases e mais de 26 mil genes. Os pesquisadores encontraram duas grandes expansões em uma família de genes - responsáveis por expressar enzimas metabólicas associadas ao citocromo P450 monoxigenase (CYP) - conhecida de participar de processos desintoxicantes. Via duplicação genética, várias cópias dos genes que expressam essas enzimas estão presentes no coala, reforçando a proteção contra efeitos tóxicos. Expansões também foram observadas em genes associados com o paladar e o olfato.
COALA Em 2018, o sequenciamento do genoma do Coala (Phascolarctos cinereus), um marsupial nativo da Austrália pertencente à família Phascolarctidae, foi publicado na Nature Genetics (Ref.35). Esses mamíferos se alimentam quase que exclusivamente da planta Eucalyptus spp., uma dieta que seria tóxica ou fatal para a maioria dos outros mamíferos. Com 16 cromossomos, o genoma dos coalas é um pouco maior do que o dos humanos, com 3,4 bilhões de pares de bases e mais de 26 mil genes. Os pesquisadores encontraram duas grandes expansões em uma família de genes - responsáveis por expressar enzimas metabólicas associadas ao citocromo P450 monoxigenase (CYP) - conhecida de participar de processos desintoxicantes. Via duplicação genética, várias cópias dos genes que expressam essas enzimas estão presentes no coala, reforçando a proteção contra efeitos tóxicos. Expansões também foram observadas em genes associados com o paladar e o olfato.
SAFRÃO Com um preço que chega a alcançar os 30 mil euros o quilo, o safrão é a mais cara especiaria no mundo, muitas vezes excedendo o preço do ouro. O safrão é colhido das flores da planta Crocus sativus, a qual floresce apenas no outono. Considerando seu alto valor, um estudo publicado na New Phytologist (Ref.37) resolveu explorar o genoma da C. sativas em busca de melhoramentos genéticos. O que se sabia é que planta é um triploide (com oito cromossomos tripletos), estéril e que não pode ser cruzada (sendo propagada apenas via linhagens clonadas). Os pesquisadores no estudo encontraram que a planta não era um híbrido entre duas espécies, como se pensava, mas um auto-triploide oriundo do cruzamento de dois indivíduos de uma mesma espécie selvagem (Crocus cartwrightianus), encontrada na Grécia, que possuíam cromossomos levemente diferentes (diferentes citotipos). Nesse caso, o material genético do gameta masculino de um dos espécimes envolvidos no cruzamento não foi reduzido durante a meiose, levando duas cópias para a fertilização do gameta feminino.
AMENDOIM O amendoim hoje cultivado (Arachis hypogaea) teve origem evolutiva na América do Sul, há cerca de 9400 anos, onde os genomas de dois ancestrais selvagens - A. duranensis e A. ipaensis - se fundiram após um evento de hibridização. Após sucessivas deleções genômicas e recombinações homólogas (fluxo de informação genética entre os dois subgenomas) ao longo do processo de domesticação, o resultado final foi um complexo genoma alotetraploide com um tamanho de 2,7 bilhões de bases.
2. Alteração de estruturas genéticas pré-existentes Novas estruturas genéticas podem ser geradas ao se modificar éxons e domínios já existentes no genoma. Nesse sentido, éxons e domínios podem ser recombinados para produzir novos genes quiméricos. Proteínas quiméricas formadas pela recombinação de genes já foram encontradas em vários organismos desde a descoberta do gene receptor LDL, incluindo lêvedos, Drosophila, Caenorhabditis elegans, mamíferos e plantas. Estima-se que esse processo de recombinação tenha contribuído com cerca de 19% dos novos éxons nos eucariotas. Novas estruturas genéticas também podem ser formadas pela cisão de genes pré-existentes. Além disso, o processo de splicing alternativo (4) entre genes duplicados pode gerar transcritos distintos que produzem RNAs não-codificantes ou polipeptídeos com funções levemente ou completamente diferentes e rapidamente alterar estruturas e funções desses genes duplicados.
3. Genes de novo Novas estruturas genéticas podem emergir a partir de sequências não-codificantes do DNA, ou seja, porções genômicas que antes não codificavam proteínas - apenas regulavam - podem passar a codificar após mutações diversas e DNA suffling (processo de diversificação genética onde pedaços de genes são misturados dando origem a novos genes). Aliás, apenas cerca de 1,5% do DNA humano codifica um produto genético (englobando 30% se as sequências de íntrons forem incluídas), ou seja, bastante matéria-prima em potencial para genes de novo. Um estudo publicado este ano na mBio (Ref.8) conseguiu identificar em uma única espécie de levedura - no caso a S. cerevisiae - 84 genes de novo espalhados entre 15 dos 16 cromossomos do seu genoma.
Já um estudo publicado no periódico Nature Ecology and Evolution (Ref.18) trouxe evidências conclusivas de que seções aleatórias e não-codificantes do DNA podem rapidamente evoluir para produzir novas proteínas. O estudo realizou análises genômicas comparativas entre 11 espécies de plantas do arroz próximo-relacionadas, incluindo a Oryza sativa, a mais comum espécie de plantio. Todas essas espécies divergiram há cerca de 3-4 milhões de anos. Os resultados das análises revelaram 175 genes de novo, onde 57% desses genes produziam novas proteínas, incluindo mais de 300 novos peptídeos. Esse achado reforça que a evolução de genes de novo é bastante comum na natureza, e indica que esse processo evolutivo promove a rápida diversificação de proteínas sob seleção positiva.
E de forma mais detalhada e mais robusta, pesquisadores em um estudo publicado em 2019 na PNAS (Ref.25), descreveram - e comprovaram experimentalmente - como peixes no Ártico - no caso, espécies da família Gadidae (conhecidas popularmente como 'bacalhau') - evoluíram um gene que codifica uma glicoproteína anti-congelante (AFGP) a partir de pequenos fragmentos não-codificantes do DNA, há cerca de 15 milhões de anos.
Através de análises comparativas do genoma de duas linhagens de bacalhau - uma que produzia a proteína AFGP e a outra que não produz -, os pesquisadores encontraram os ancestrais genéticos do gene anti-congelamento em uma região de DNA não-codificante. Em seguida, os pesquisadores mostraram como o gene foi formado, passo a passo, em um processo que envolveu vários eventos moleculares, sendo o principal deles múltiplas duplicações de um pequeno segmento genômico constituído de apenas 9 nucleotídeos, criando uma mais longa sequência de repetições. Esse evento específico de duplicações deu origem a uma sequência que, se traduzida, codifica uma cadeia repetitiva de uma série com três aminoácidos (treonina-alanina-alanina), a qual possui a capacidade química de se ligar a cristais de gelo no sangue e prevenir o perigoso crescimento de cristais. Nesse sentido, a nova sequência genética passou por subsequentes eventos evolutivos - incluindo deleções e uma translocação - que levaram à evolução de um gene completo, e capaz de ser transcrito em RNA e este traduzido (o passo final foi a aquisição do elemento regulatório promotor via translocação). Uma vez gerado o gene anti-congelante, sua proteína expressa garante maior capacidade de sobrevivência para esses peixes (vantagem adaptativa) nos mares gelados do extremo Norte, sendo portanto conservado ao longo das gerações (fixado) e eventuais especiações.
Por fim, em um estudo publicado no periódico mBio (Ref.39) pesquisadores experimentalmente demonstraram a emergência de novas proteínas adaptativas a partir de sequências de DNA aleatórias não-funcionais. No caso, utilizando uma biblioteca com 500 milhões de sequências genéticas aleatórias capazes de expressarem peptídeos com funções biológicas, os pesquisadores escolheram várias dessas sequências - sem critério de preferência - e as inseriram no plasmídeo de bactérias E. coli. Eventualmente eles identificaram diversos peptídeos de cadeira curta (22-25 aminoácidos de comprimento) que estavam sendo selecionados nas populações bacterianas por conferirem resistência a uma importante classe de antibióticos (aminoglicosídeos). Esses peptídeos eram altamente hidrofóbicos e, ao se anexarem na membrana plasmática das bactérias, diminuíam o potencial de próton nessa estrutura, conferindo resistência até 48 vezes maior do que antes encontrada nas populações originais. Em outras palavras, foi demonstrado a evolução de novos adaptativos a partir de sequências aleatórias inseridas no material genético dessas bactérias, o que dá suporte experimental para a origem de novos genes via reciclagem de sequências não-codificantes (De novo gene).
4. Transferência lateral de genes (TLG) Também conhecida como transferência horizontal de genes (THG), esse processo está massivamente presente nos seres procariotas, onde genes são compartilhados entre genomas de táxons distintos. Genes de resistência a antibióticos são geralmente adquiridos por várias bactérias a partir desse processo de compartilhamento genético. No geral, quaisquer genes livres no ambiente, de origens diversas, podem ser capturados e incorporados pelos procariontes via transferência lateral. A TLG é muito importante na evolução dos seres procariontes (incluindo Archaea) (Ref.46), mas também já foi reportada em seres eucariontes, como plantas, insetos e fungos. Basicamente, genes de espécies distintas são capturados (5) e adicionados ao genoma de outras espécies, trazendo novidades fenotípicas e acumulando informação genética. Aliás, é impossível descrever a evolução de comunidades microbianas sem levar a TLG em conta (Ref.47).
A TLG pode ocorrer por três principais mecanismos (no caso, entre bactérias):
- Transformação: Envolve a incorporação de fragmentos curtos de puro DNA por bactérias naturalmente transformáveis;
- Transdução: Envolve transferência de DNA de uma bactéria para outra via bacteriófagos (vírus que atacam bactérias);
- Conjugação: Envolve transferência de DNA via pelos sexuais e requer contato direto entre as células bacterianas.
RESISTÊNCIA BACTERIANA Muitos dos genes de resistência a antibióticos nas bactérias são carregados em plasmídeos, transpósons e integrões que podem agir como vetores de transferência desses genes para outros membros da mesma espécie de bactéria para outras espécies ou gêneros. Assim, esses microrganismos não precisam necessariamente ter que esperar por uma mutação aleatória emergir em um indivíduo de uma população específica para se adaptar a um novo antibiótico X. Se outra espécie ou cepa já passou pelo processo de acumular as mutações necessárias para resistir ao medicamento, os genes de resistência provavelmente já estão em fluxo entre as populações bacterianas. Isso significa que uma população que encontra o antibiótico X pela primeira vez pode já ter alguns indivíduos carregando genes de resistência adquiridos de fora, permitindo que essa população evolua rápido contra o medicamento.
Por muito tempo persistiu a controversa questão sobre se a TLG também atua no sentido 'procarionte → eucarionte', ou se apenas os procariontes conseguem reaproveitar genes horizontalmente transferidos, seja de outros procariontes seja de eucariontes. Mas estudos nos últimos anos (!) têm deixado claro que a TLG entre procariontes e eucariontes ocorre nos dois sentidos e parece ter impactado de forma importante a história evolutiva eucariótica. A TLG ocorre inclusive entre eucariontes. Notavelmente, vários genes transferidos horizontalmente de bactérias, fungos e vírus para plantas parecem ter sido cruciais para a colonização terrestre de antigas plantas (Ref.49-50). Em um estudo publicado em 2021 no periódico New Phytologist (Ref.51), pesquisadores mostraram amplos processos de TLG entre espécies distintas de gramíneas, com transferência de genes funcionais tanto para espécies selvagens quanto para espécies domesticadas e através de mecanismos ainda não muito bem esclarecidos.
TRANSFERÊNCIA LATERAL DE GENES VIRAIS
Enquanto desconhecemos a real extensão das transferências laterais de genes entre procariontes e eucariontes, os vírus - em específico os retrovírus no caso dos vertebrados - promovem massivas transferências de material genético para eucariontes diversos, impactando de forma crucial a evolução desses organismos (Ref.48).
Infecções virais de células germinativas (gametas ou células da fase inicial do desenvolvimento embrionário, por exemplo) podem levar a genes ou genomas virais a se tornarem integrados aos cromossomos do hospedeiros e serem passíveis de transmissão entre gerações como novos alelos. Essas inserções, conhecidas como EVE (elementos endógenos virais), são geralmente eliminadas do gene pool do hospedeiro dentro de um pequeno número de gerações. No entanto, os EVE também podem aumentar em frequência, e alguns eventualmente alcançam fixação (100% de frequência em uma dada população). Os EVE no genoma de eucariontes podem representar fragmentos de genoma viral ou o genoma viral inteiro (pró-vírus). Sequências completas podem levar a manifestação da doença virótica nos descendentes do hospedeiro infectado, ou subsequentes mutações podem torná-las inativas.
Nos genomas dos animais, incluindo humanos, a maioria dos EVE são derivados de vírus de transcrição reversa (rtRNA), como os retrovírus (5). Retrovírus são os únicos vírus de animais que integram seu material genético no genoma das células hospedeiras como um passo obrigatório na sua estratégia de replicação, e, portanto, predispõem a formação de EVE. Nesse sentido, os retrovírus são, de longe, as maiores fontes de EVE.
Os retrovírus endógenos, aliás, são importantes fatores agindo na evolução genômica dos vertebrados, não apenas como parasitas genéticos, mas também ao introduzirem novidades genéticas úteis no material genético dos hospedeiros. Alguns cientistas também propõem mecanismos onde os vírus podem auxiliar a ocorrência de TLG não-viral nos eucariontes.
Aliás, os pesquisadores hoje estudam antigos ancestrais de vírus na Paleovirologia sequenciando o genoma de espécies diversas em reinos diversos na busca por EVE, as quais podem ser conservadas por vários milhões de anos após serem fixadas no genoma do hospedeiro ancestral. Sequências completas de genoma viral são mais raras na forma de EVE, mas fragmentos são amplamente espalhados na natureza. Já foram identificados EVE em enorme abundância nas plantas, animais e fungos, e uma considerável porção dos genomas de vertebrados é constituída de EVE.. Para se ter uma ideia, cerca de 8% do genoma humano é composto por EVE. Isso é superior a 6 vezes mais DNA do que é encontrado em todos os nossos ~20 mil genes codificadores de proteínas! Os retrovírus endógenos parecem ter se inserido no DNA humano em diferentes pontos da história evolutiva, variando desde 100 milhões de anos atrás até talvez menos de 100 mil anos atrás.
Importante também lembrar que os vírus, assim como os procariontes, conseguem incorporar RNA ou DNA do ambiente - especificamente de hospedeiros -, fomentando a evolução desses seres. Nesse sentido...
TL MEDIADA POR VÍRUS
Desde a década de 1970 já é sabido que genes de hospedeiros são frequentemente capturados por vírus e integrados ao genoma viral. Aliás, uma substancial fração dos genes codificados por grandes vírus dsDNA (até 30% em alguns vírus da herpes) têm origem de eucariontes. Esses genes se encontram fixados em várias espécies de vírus, provavelmente devido a vantagens adaptativas. Nesse sentido, pesquisadores têm proposto que uma forma comum de transferência lateral entre eucariontes pode estar sendo mediada por vírus. Esse fluxo genético, portanto, requereria:
i) aquisição de fragmentos do genoma hospedeiro por um vírus;
ii) integração desse fragmento associado ao genoma viral ao genoma de outro hospedeiro.
EXEMPLO NOTÁVEL
Recentemente o genoma do tubarão-branco (Carcharodon carcharias) foi inteiramente sequenciado e descrito em uma publicação no periódico PNAS (Ref.36), revelando um tamanho genômico de 4,63 Gb (~1,5X o genoma humano), 24520 genes, 58,5% de sequências repetidas e a presença de uma enorme quantidade de transposons, especificamente do tipo LINE. É a mais alta proporção de LINEs (quase 30%) descoberta em vertebrados até o momento. Esse tipo de transposon - particularmente o LINE-1 - causa instabilidade genômica ao criar quebras de cadeia dupla no DNA, o que pode ter fomentado a evolução de vários mecanismos eficientes de reparação genética e de cura de feridas que são notáveis nesses predadores. No tubarão-branco, o LINE mais prevalente é o LINE-3/CR1, o qual parece possuir as mesmas capacidades de retro-transposição do LINE-1.
- Continua após o anúncio -
CONCLUSÃO
Existem vários mecanismos moleculares para a produção, deleção e acúmulo de novos genes, os quais baseiam e fomentam o processo evolutivo, e esses mecanismos frequentemente cooperam entre si para as transformações genômicas. Para o acúmulo de informações genéticas e consequente expansão genômica, a duplicação genética (mutação), a transposição de elementos e a transferência lateral de genes (compartilhamento genético) são os mais importantes desses mecanismos. Os padrões no material genético dos seres vivos não pode ser explicado fora de um contexto evolutivo.
REFERÊNCIAS CIENTÍFICAS
ÓPIO Publicado na Science (Ref.33), o sequenciamento do genoma do ópio (Papaver somnifeum L.) revelou 2,72 Gb distribuídas em 11 cromossomos, e uma assinatura de duplicação genômica inteira há cerca de 7,8 milhões de anos, e uma provável duplicação (ões) genética (s) há cerca de 100 milhões de anos durante a divergência das famílias Papaveraceae e Ranunculaceae. No estudo, os pesquisadores mostraram como duplicações, rearranjamentos e fusões de genes levaram à evolução de produtos metabólitos especializados na papoula do ópio, como a morfina.
 COALA Em 2018, o sequenciamento do genoma do Coala (Phascolarctos cinereus), um marsupial nativo da Austrália pertencente à família Phascolarctidae, foi publicado na Nature Genetics (Ref.35). Esses mamíferos se alimentam quase que exclusivamente da planta Eucalyptus spp., uma dieta que seria tóxica ou fatal para a maioria dos outros mamíferos. Com 16 cromossomos, o genoma dos coalas é um pouco maior do que o dos humanos, com 3,4 bilhões de pares de bases e mais de 26 mil genes. Os pesquisadores encontraram duas grandes expansões em uma família de genes - responsáveis por expressar enzimas metabólicas associadas ao citocromo P450 monoxigenase (CYP) - conhecida de participar de processos desintoxicantes. Via duplicação genética, várias cópias dos genes que expressam essas enzimas estão presentes no coala, reforçando a proteção contra efeitos tóxicos. Expansões também foram observadas em genes associados com o paladar e o olfato.
COALA Em 2018, o sequenciamento do genoma do Coala (Phascolarctos cinereus), um marsupial nativo da Austrália pertencente à família Phascolarctidae, foi publicado na Nature Genetics (Ref.35). Esses mamíferos se alimentam quase que exclusivamente da planta Eucalyptus spp., uma dieta que seria tóxica ou fatal para a maioria dos outros mamíferos. Com 16 cromossomos, o genoma dos coalas é um pouco maior do que o dos humanos, com 3,4 bilhões de pares de bases e mais de 26 mil genes. Os pesquisadores encontraram duas grandes expansões em uma família de genes - responsáveis por expressar enzimas metabólicas associadas ao citocromo P450 monoxigenase (CYP) - conhecida de participar de processos desintoxicantes. Via duplicação genética, várias cópias dos genes que expressam essas enzimas estão presentes no coala, reforçando a proteção contra efeitos tóxicos. Expansões também foram observadas em genes associados com o paladar e o olfato.SAFRÃO Com um preço que chega a alcançar os 30 mil euros o quilo, o safrão é a mais cara especiaria no mundo, muitas vezes excedendo o preço do ouro. O safrão é colhido das flores da planta Crocus sativus, a qual floresce apenas no outono. Considerando seu alto valor, um estudo publicado na New Phytologist (Ref.37) resolveu explorar o genoma da C. sativas em busca de melhoramentos genéticos. O que se sabia é que planta é um triploide (com oito cromossomos tripletos), estéril e que não pode ser cruzada (sendo propagada apenas via linhagens clonadas). Os pesquisadores no estudo encontraram que a planta não era um híbrido entre duas espécies, como se pensava, mas um auto-triploide oriundo do cruzamento de dois indivíduos de uma mesma espécie selvagem (Crocus cartwrightianus), encontrada na Grécia, que possuíam cromossomos levemente diferentes (diferentes citotipos). Nesse caso, o material genético do gameta masculino de um dos espécimes envolvidos no cruzamento não foi reduzido durante a meiose, levando duas cópias para a fertilização do gameta feminino.
AMENDOIM O amendoim hoje cultivado (Arachis hypogaea) teve origem evolutiva na América do Sul, há cerca de 9400 anos, onde os genomas de dois ancestrais selvagens - A. duranensis e A. ipaensis - se fundiram após um evento de hibridização. Após sucessivas deleções genômicas e recombinações homólogas (fluxo de informação genética entre os dois subgenomas) ao longo do processo de domesticação, o resultado final foi um complexo genoma alotetraploide com um tamanho de 2,7 bilhões de bases.
BANANA Cultivares de banana como o conhecemos hoje - um fruto sem sementes - é o resultado de hibridização entre espécies e subespécies do gênero Musa que divergiram em diferentes regiões e ilhas tropicais do Sudeste Asiático e Melanésia Ocidental (Ref.40-41). Em específico, a espécie Musa acuminata (2n = 22) está envolvida com a origem de todos os cultivares de banana, com exceção da banana Fe'i rica em vitaminas. Entre outras espécies envolvidas nos processos de hibridização temos a M. textilis, M. schizocarpa e M. balbisiana (2n = 22), essa última associada com a M. acuminata em vários cultivares. A M. acuminata é dividida em várias subespécies distribuídas desde o nordeste da Índia até a Nova Guiné, incluindo: siamea, burmannica, burmannicoïdes, malaccensis, truncata, errans, microcarpa, zebrina e banksii. Os sucessivos processos de hibridização e introgressão produziram diversos híbridos diploides e triploides, sendo selecionados para frutos partenocárpicos (desenvolvidos sem fertilização) e estéreis, e, consequentemente, sem sementes. As plantas selecionadas - e inférteis - foram então sendo propagadas vegetativamente por séculos ou mesmo milênios.
-----------
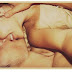
ALERTA: Além de serem pouco comestíveis, o consumo de bananas selvagens deve ser evitado. A ingestão das sementes nos frutos selvagens pode causar sérios quadros de obstrução intestinal devido à formação de fitobezoares, e relatos de caso nesse sentido têm sido ocasionalmente reportados na literatura acadêmica (Ref.42).
 |
| Fitobezoares derivados da ingestão de bananas selvagens da espécie Musa acuminata, removidos do intestino de um paciente (diâmetro máximo de 5 cm). Slesak et al., 2011 |
2. Alteração de estruturas genéticas pré-existentes Novas estruturas genéticas podem ser geradas ao se modificar éxons e domínios já existentes no genoma. Nesse sentido, éxons e domínios podem ser recombinados para produzir novos genes quiméricos. Proteínas quiméricas formadas pela recombinação de genes já foram encontradas em vários organismos desde a descoberta do gene receptor LDL, incluindo lêvedos, Drosophila, Caenorhabditis elegans, mamíferos e plantas. Estima-se que esse processo de recombinação tenha contribuído com cerca de 19% dos novos éxons nos eucariotas. Novas estruturas genéticas também podem ser formadas pela cisão de genes pré-existentes. Além disso, o processo de splicing alternativo (4) entre genes duplicados pode gerar transcritos distintos que produzem RNAs não-codificantes ou polipeptídeos com funções levemente ou completamente diferentes e rapidamente alterar estruturas e funções desses genes duplicados.
- (4) Para saber mais, acesse: Splicing pode ser a chave para explicar o rápido processo evolutivo de domesticação dos girassóis
3. Genes de novo Novas estruturas genéticas podem emergir a partir de sequências não-codificantes do DNA, ou seja, porções genômicas que antes não codificavam proteínas - apenas regulavam - podem passar a codificar após mutações diversas e DNA suffling (processo de diversificação genética onde pedaços de genes são misturados dando origem a novos genes). Aliás, apenas cerca de 1,5% do DNA humano codifica um produto genético (englobando 30% se as sequências de íntrons forem incluídas), ou seja, bastante matéria-prima em potencial para genes de novo. Um estudo publicado este ano na mBio (Ref.8) conseguiu identificar em uma única espécie de levedura - no caso a S. cerevisiae - 84 genes de novo espalhados entre 15 dos 16 cromossomos do seu genoma.
Já um estudo publicado no periódico Nature Ecology and Evolution (Ref.18) trouxe evidências conclusivas de que seções aleatórias e não-codificantes do DNA podem rapidamente evoluir para produzir novas proteínas. O estudo realizou análises genômicas comparativas entre 11 espécies de plantas do arroz próximo-relacionadas, incluindo a Oryza sativa, a mais comum espécie de plantio. Todas essas espécies divergiram há cerca de 3-4 milhões de anos. Os resultados das análises revelaram 175 genes de novo, onde 57% desses genes produziam novas proteínas, incluindo mais de 300 novos peptídeos. Esse achado reforça que a evolução de genes de novo é bastante comum na natureza, e indica que esse processo evolutivo promove a rápida diversificação de proteínas sob seleção positiva.
E de forma mais detalhada e mais robusta, pesquisadores em um estudo publicado em 2019 na PNAS (Ref.25), descreveram - e comprovaram experimentalmente - como peixes no Ártico - no caso, espécies da família Gadidae (conhecidas popularmente como 'bacalhau') - evoluíram um gene que codifica uma glicoproteína anti-congelante (AFGP) a partir de pequenos fragmentos não-codificantes do DNA, há cerca de 15 milhões de anos.
Através de análises comparativas do genoma de duas linhagens de bacalhau - uma que produzia a proteína AFGP e a outra que não produz -, os pesquisadores encontraram os ancestrais genéticos do gene anti-congelamento em uma região de DNA não-codificante. Em seguida, os pesquisadores mostraram como o gene foi formado, passo a passo, em um processo que envolveu vários eventos moleculares, sendo o principal deles múltiplas duplicações de um pequeno segmento genômico constituído de apenas 9 nucleotídeos, criando uma mais longa sequência de repetições. Esse evento específico de duplicações deu origem a uma sequência que, se traduzida, codifica uma cadeia repetitiva de uma série com três aminoácidos (treonina-alanina-alanina), a qual possui a capacidade química de se ligar a cristais de gelo no sangue e prevenir o perigoso crescimento de cristais. Nesse sentido, a nova sequência genética passou por subsequentes eventos evolutivos - incluindo deleções e uma translocação - que levaram à evolução de um gene completo, e capaz de ser transcrito em RNA e este traduzido (o passo final foi a aquisição do elemento regulatório promotor via translocação). Uma vez gerado o gene anti-congelante, sua proteína expressa garante maior capacidade de sobrevivência para esses peixes (vantagem adaptativa) nos mares gelados do extremo Norte, sendo portanto conservado ao longo das gerações (fixado) e eventuais especiações.
Por fim, em um estudo publicado no periódico mBio (Ref.39) pesquisadores experimentalmente demonstraram a emergência de novas proteínas adaptativas a partir de sequências de DNA aleatórias não-funcionais. No caso, utilizando uma biblioteca com 500 milhões de sequências genéticas aleatórias capazes de expressarem peptídeos com funções biológicas, os pesquisadores escolheram várias dessas sequências - sem critério de preferência - e as inseriram no plasmídeo de bactérias E. coli. Eventualmente eles identificaram diversos peptídeos de cadeira curta (22-25 aminoácidos de comprimento) que estavam sendo selecionados nas populações bacterianas por conferirem resistência a uma importante classe de antibióticos (aminoglicosídeos). Esses peptídeos eram altamente hidrofóbicos e, ao se anexarem na membrana plasmática das bactérias, diminuíam o potencial de próton nessa estrutura, conferindo resistência até 48 vezes maior do que antes encontrada nas populações originais. Em outras palavras, foi demonstrado a evolução de novos adaptativos a partir de sequências aleatórias inseridas no material genético dessas bactérias, o que dá suporte experimental para a origem de novos genes via reciclagem de sequências não-codificantes (De novo gene).
4. Transferência lateral de genes (TLG) Também conhecida como transferência horizontal de genes (THG), esse processo está massivamente presente nos seres procariotas, onde genes são compartilhados entre genomas de táxons distintos. Genes de resistência a antibióticos são geralmente adquiridos por várias bactérias a partir desse processo de compartilhamento genético. No geral, quaisquer genes livres no ambiente, de origens diversas, podem ser capturados e incorporados pelos procariontes via transferência lateral. A TLG é muito importante na evolução dos seres procariontes (incluindo Archaea) (Ref.46), mas também já foi reportada em seres eucariontes, como plantas, insetos e fungos. Basicamente, genes de espécies distintas são capturados (5) e adicionados ao genoma de outras espécies, trazendo novidades fenotípicas e acumulando informação genética. Aliás, é impossível descrever a evolução de comunidades microbianas sem levar a TLG em conta (Ref.47).
- (5) Leitura recomendada: Cientistas, pela primeira vez, flagram uma bactéria capturando um pedaço de DNA para acelerar sua evolução
A TLG pode ocorrer por três principais mecanismos (no caso, entre bactérias):
- Transformação: Envolve a incorporação de fragmentos curtos de puro DNA por bactérias naturalmente transformáveis;
- Transdução: Envolve transferência de DNA de uma bactéria para outra via bacteriófagos (vírus que atacam bactérias);
- Conjugação: Envolve transferência de DNA via pelos sexuais e requer contato direto entre as células bacterianas.
RESISTÊNCIA BACTERIANA Muitos dos genes de resistência a antibióticos nas bactérias são carregados em plasmídeos, transpósons e integrões que podem agir como vetores de transferência desses genes para outros membros da mesma espécie de bactéria para outras espécies ou gêneros. Assim, esses microrganismos não precisam necessariamente ter que esperar por uma mutação aleatória emergir em um indivíduo de uma população específica para se adaptar a um novo antibiótico X. Se outra espécie ou cepa já passou pelo processo de acumular as mutações necessárias para resistir ao medicamento, os genes de resistência provavelmente já estão em fluxo entre as populações bacterianas. Isso significa que uma população que encontra o antibiótico X pela primeira vez pode já ter alguns indivíduos carregando genes de resistência adquiridos de fora, permitindo que essa população evolua rápido contra o medicamento.
------------
> Em um estudo publicado recentemente no periódico Cell (Ref.52), pesquisadores descreveram um novo mecanismo de TLG em procariontes. No caso, os pesquisadores analisaram uma bactéria fotossintética marinha (cianobactérias) do gênero Prochlorococcus, e mostraram que esses microrganismos transferem blocos inteiros de genes entre si - com funções diversas desde metabolismo de nutrientes até defesa contra vírus - mesmo quando estão separadas por grandes distâncias. O mecanismo envolve agentes genéticos móveis previamente desconhecidos e apelidados no novo estudo de "tycheposons", os quais podem desprender sequências com vários genes do DNA das bactérias; esses blocos genéticos desprendidos podem então ser transportados na água para outros indivíduos por um ou outro sistema de transporte, incluindo vesículas derivadas da membrana celular do próprio doador. O fenômeno parece ocorrer também em várias outras bactérias marinhas.
------------
Por muito tempo persistiu a controversa questão sobre se a TLG também atua no sentido 'procarionte → eucarionte', ou se apenas os procariontes conseguem reaproveitar genes horizontalmente transferidos, seja de outros procariontes seja de eucariontes. Mas estudos nos últimos anos (!) têm deixado claro que a TLG entre procariontes e eucariontes ocorre nos dois sentidos e parece ter impactado de forma importante a história evolutiva eucariótica. A TLG ocorre inclusive entre eucariontes. Notavelmente, vários genes transferidos horizontalmente de bactérias, fungos e vírus para plantas parecem ter sido cruciais para a colonização terrestre de antigas plantas (Ref.49-50). Em um estudo publicado em 2021 no periódico New Phytologist (Ref.51), pesquisadores mostraram amplos processos de TLG entre espécies distintas de gramíneas, com transferência de genes funcionais tanto para espécies selvagens quanto para espécies domesticadas e através de mecanismos ainda não muito bem esclarecidos.
(!) Sugestão de leitura:
- Transferência lateral de genes apontada em eucariontes
- Gene de uma antiga bactéria ajuda os carrapatos a espalharem a doença de Lyme
- Inseto roubou gene de planta para se proteger de tóxicos compostos planta-específicos
- Cientistas descobrem a origem evolucionária de um componente essencial do olho humano
TRANSFERÊNCIA LATERAL DE GENES VIRAIS
Enquanto desconhecemos a real extensão das transferências laterais de genes entre procariontes e eucariontes, os vírus - em específico os retrovírus no caso dos vertebrados - promovem massivas transferências de material genético para eucariontes diversos, impactando de forma crucial a evolução desses organismos (Ref.48).
Infecções virais de células germinativas (gametas ou células da fase inicial do desenvolvimento embrionário, por exemplo) podem levar a genes ou genomas virais a se tornarem integrados aos cromossomos do hospedeiros e serem passíveis de transmissão entre gerações como novos alelos. Essas inserções, conhecidas como EVE (elementos endógenos virais), são geralmente eliminadas do gene pool do hospedeiro dentro de um pequeno número de gerações. No entanto, os EVE também podem aumentar em frequência, e alguns eventualmente alcançam fixação (100% de frequência em uma dada população). Os EVE no genoma de eucariontes podem representar fragmentos de genoma viral ou o genoma viral inteiro (pró-vírus). Sequências completas podem levar a manifestação da doença virótica nos descendentes do hospedeiro infectado, ou subsequentes mutações podem torná-las inativas.
Nos genomas dos animais, incluindo humanos, a maioria dos EVE são derivados de vírus de transcrição reversa (rtRNA), como os retrovírus (5). Retrovírus são os únicos vírus de animais que integram seu material genético no genoma das células hospedeiras como um passo obrigatório na sua estratégia de replicação, e, portanto, predispõem a formação de EVE. Nesse sentido, os retrovírus são, de longe, as maiores fontes de EVE.
- (5) Leitura recomendada: HIV indetectável é intransmissível
Os retrovírus endógenos, aliás, são importantes fatores agindo na evolução genômica dos vertebrados, não apenas como parasitas genéticos, mas também ao introduzirem novidades genéticas úteis no material genético dos hospedeiros. Alguns cientistas também propõem mecanismos onde os vírus podem auxiliar a ocorrência de TLG não-viral nos eucariontes.
Aliás, os pesquisadores hoje estudam antigos ancestrais de vírus na Paleovirologia sequenciando o genoma de espécies diversas em reinos diversos na busca por EVE, as quais podem ser conservadas por vários milhões de anos após serem fixadas no genoma do hospedeiro ancestral. Sequências completas de genoma viral são mais raras na forma de EVE, mas fragmentos são amplamente espalhados na natureza. Já foram identificados EVE em enorme abundância nas plantas, animais e fungos, e uma considerável porção dos genomas de vertebrados é constituída de EVE.. Para se ter uma ideia, cerca de 8% do genoma humano é composto por EVE. Isso é superior a 6 vezes mais DNA do que é encontrado em todos os nossos ~20 mil genes codificadores de proteínas! Os retrovírus endógenos parecem ter se inserido no DNA humano em diferentes pontos da história evolutiva, variando desde 100 milhões de anos atrás até talvez menos de 100 mil anos atrás.
Aliás, genes humanos cruciais para o desenvolvimento da placenta são EVE! São pelo menos oito genes conhecidos como sincitinas (ERVW1, ERVFRD-1, ERVV-1, ERVV-2, ERVH48-1, ERVMER34-1, ERV3-1 e ERVK13-1) e um gene conhecido como Suppressyn (SUPYN) derivados de antigas sequências virais integradas no nosso DNA. Esses genes são expressos (no embrião em desenvolvimento) desde a fase de implantação até o final da gestação, e são responsáveis por promover ou inibir (!) fusões celulares críticas para o desenvolvimento placentário. Aliás, sincitinas parecem ser expressas em todos os mamíferos com placenta, mas não necessariamente derivadas das mesmas sequências retrovirais (!). E vários outros EVE no nosso genoma parecem ter funções importantes ou possuem algum tipo de papel nos tecidos humanos, incluindo proteção antiviral (Ref.44). Por outro lado, sequências retrovirais integradas no nosso DNA também estão associadas ao desenvolvimento de doenças diversas, incluindo câncer e desordens neurodegenerativas (Ref.44).
------------
(!) Por exemplo, o gene ERVH48-1 (também conhecido como Suppressyn ou SUPYN) atua inibindo ao invés de promover fusões dos trofoblastos, enquanto as principais sincitinas humanas (ERVWI ou sincitina 1, e ERVFRD-1 ou sincitina-2) atuam promovendo fusões celulares. A integração das sequências genéticas retrovirais ancestrais e a emergência desses genes ocorreram em diferentes períodos ao longo do processo evolutivo dos primatas. Por exemplo, o ERVW-1 emergiu há 20-30 milhões de anos, e está ausente nos macacos do Novo Mundo. Já o ERVRD-1 foi integrado aproximadamente 20 milhões de anos prévios à emergência do ERVW-1, e está presente em todos os primatas, com exceção dos prossímios. Além do desenvolvimento placentário, esses genes também parecem proteger os fetos de infecções virais (Ref.45). Para uma revisão recente sobre esses genes, acesse a Ref.43.
------------
Importante também lembrar que os vírus, assim como os procariontes, conseguem incorporar RNA ou DNA do ambiente - especificamente de hospedeiros -, fomentando a evolução desses seres. Nesse sentido...
TL MEDIADA POR VÍRUS
Desde a década de 1970 já é sabido que genes de hospedeiros são frequentemente capturados por vírus e integrados ao genoma viral. Aliás, uma substancial fração dos genes codificados por grandes vírus dsDNA (até 30% em alguns vírus da herpes) têm origem de eucariontes. Esses genes se encontram fixados em várias espécies de vírus, provavelmente devido a vantagens adaptativas. Nesse sentido, pesquisadores têm proposto que uma forma comum de transferência lateral entre eucariontes pode estar sendo mediada por vírus. Esse fluxo genético, portanto, requereria:
i) aquisição de fragmentos do genoma hospedeiro por um vírus;
ii) integração desse fragmento associado ao genoma viral ao genoma de outro hospedeiro.
- (6) Leitura recomendada: O que é a deriva genética?
5. Elementos transponíveis Elementos repetidos do DNA englobam uma grande fração do genoma de eucariotas (na parte não-codificante do DNA), e alguns desses elementos - sequências genéticas - são capazes de se moverem dentro do genoma (transposons e retrotransposons). Essa movimentação ocorre via um mecanismo de corta-e-cola, e são uma importante força promovendo mudanças genéticas e, consequentemente, atuam de forma importante na evolução de muitos genomas.
No geral, os chamados elementos transponíveis (Transposables Elements, TEs) têm sido identificados em todos os organismos, procarióticos e eucarióticos, e podem ocupar uma alta proporção do genoma de várias espécies. Por exemplo, os ETs chegam a englobar cerca de 10% do genoma de vários peixes, 37% do genoma de ratos, 45% do genoma humano, e acima de 80% do genoma de algumas plantas, como o milho. De bactérias até humanos, os TEs vêm se acumulando ao longo do tempo e continuam a moldar os genomas através das suas mobilizações. Os TEs são capazes de produzir várias alterações genéticas via inserção como uma consequência do processo de transposição (inserções, excisões, duplicações ou translocações nos locais de integração). Por exemplo, transposons pode inativar ou alterar a expressão de genes via inserção dentro de íntrons, éxons ou regiões regulatórias. TEs podem participar também da reorganização do genoma pela mobilização de DNA não-transposon ou agindo como substratos de recombinação (o qual ocorre entre duas sequências de transponson localizadas no mesmo ou diferentes cromossomos), e também servirem de matéria-prima para a formação de novos genes e RNA não-codificantes. Nesse sentido, TEs podem participar da perda (redução) ou ganho (expansão) de DNA genômico - revertendo ou criando mutações -, e, portanto, alterando o genótipo celular.
Existem também os transposons de RNA, os quais funcionam via transcrição reversa de um intermediário RNA (mecanismo replicativo). Esses transposons de RNA podem ter a presença de Repetições Terminais Longas (Long Terminal Repeats, LTR) flanqueando o corpo principal do retroelemento, e são similares em estrutura e ciclo de vida aos retrovírus. Aliás, os transposons de RNA LTR englobam os já mencionados retrovírus endógenos.
Na prática, os TEs, como regra, são DNAs parasitas cuja única função é replicar e propagar a si mesmos, e inclusive passam de genoma para genoma via transferência lateral de genes. Nesse sentido, o genoma pode, em última análise, ser visto como um ecossistema habitado por diversas comunidades de TEs, os quais procuram se propagar e se multiplicar através de sofisticadas interações entre si e com outros componentes das células hospedeiras. Essas interações englobam processos como parasitismo, cooperação e competição, e raramente os TEs se encontram distribuídos aleatoriamente nos genomas.
É válido também lembrar que os transposons de DNA, apesar de constituírem grande parte do DNA humano, atualmente não se encontram móveis no nosso genoma, mas estavam ativos durante a evolução dos primeiros primatas até cerca de 37 milhões de anos atrás. No entanto, os transposons de RNA incluem elementos ativos e inativos, e estes provavelmente estão relacionados com a evolução da linhagem dos homininis a partir do ancestral comum que compartilhamos com os bonobos e chimpanzés. Apesar das transposições em células somáticas serem limitadas, existem mais do que suficientes evidências de que TEs estão ativos nessas células em vários organismos. Nas células somáticas humanas, as transposições parecem ocorrer em significativa extensão nos neurônios e em células cancerígenas.
Já foi demonstrado também que variações genéticas causadas pela expansão de TEs podem ajudar espécies a se adaptarem rapidamente a novos habitats (Ref.34), potencialmente explicando o Paradoxo de Invasão, onde espécies invasoras conseguem sucesso mesmo sofrendo robusto processo de deriva genética - via efeito fundador - associado a uma redução no potencial adaptativo e evolucionário (menor variabilidade genética intra-população).
Algumas respostas moleculares (ex: taxa de transcrição) podem também ser moduladas por elementos transponíveis, levando a respostas plásticas. Por exemplo, exposição de moscas do gênero Drosophila (pertencentes à linhagem Oregon R) a um agente pró-oxidante por 5 geração já mostrou resultar em uma elevada atividade transcricional em ambos os elementos transponíveis teloméricos e não-teloméricos, estendendo o comprimento telomérico desses insetos (Ref.38). Dessa forma, os elementos transponíveis podem participar também do processo evolutivo via plasticidade fenotípica.
Já foi demonstrado também que variações genéticas causadas pela expansão de TEs podem ajudar espécies a se adaptarem rapidamente a novos habitats (Ref.34), potencialmente explicando o Paradoxo de Invasão, onde espécies invasoras conseguem sucesso mesmo sofrendo robusto processo de deriva genética - via efeito fundador - associado a uma redução no potencial adaptativo e evolucionário (menor variabilidade genética intra-população).
Algumas respostas moleculares (ex: taxa de transcrição) podem também ser moduladas por elementos transponíveis, levando a respostas plásticas. Por exemplo, exposição de moscas do gênero Drosophila (pertencentes à linhagem Oregon R) a um agente pró-oxidante por 5 geração já mostrou resultar em uma elevada atividade transcricional em ambos os elementos transponíveis teloméricos e não-teloméricos, estendendo o comprimento telomérico desses insetos (Ref.38). Dessa forma, os elementos transponíveis podem participar também do processo evolutivo via plasticidade fenotípica.
MECANISMOS DE TRANSPOSIÇÃO
Os transposons de DNA consistem de um gene transposase que é flanqueado por duas Repetições Terminais Invertidas (Terminal Inverted Repeats, TIRs). A enzima transposase expressa reconhece esses TIRs para realizar a excisão do corpo do transposon de DNA, o qual é inserido em uma nova localização genômica. Na inserção, o local alvo do DNA é duplicado, resultando em Target Site Duplications (TSDs).
Os TEs são divididos em famílias. Os elementos da superfamília Tc1/mariner são provavelmente os mais amplamente distribuídos na natureza, representados em seres como otíferos, fungos, plantas, peixes e mamíferos. Os elementos Tc1/mariner possuem entre 1 e 5 quilobases (kb) - cada quilobase representa mil bases de nucleotídeos/letras do DNA - em comprimento, e codificam uma transposase de 282 a 345 aminoácidos a qual é flanqueada por dois TIRs que variam entre 17 e 1100 bp (par de bases) de comprimento.
MECANISMOS REGULATÓRIOS
É evidente também que essa movimentação de sequências genéticas de um lado para o outro, e a expressão de proteínas não associadas aos genes específicos do hospedeiro, eleva o risco de efeitos deletérios no DNA - instabilidade genética e disposição ao câncer, por exemplo -, e várias doenças estão relacionadas aos transposons. Por isso o genoma dos organismos evoluíram mecanismos de regulação e de controle das transposições. Da mesma forma, os transposons também evoluem mecanismos de defesa contra a repressão do genoma do hospedeiro*.
Aliás, os elementos transponíveis podem até mesmo serem domesticados para executarem funções úteis para a célula, e vários exemplos já foram descritos na literatura acadêmica (Ref.20-22). Um estudo publicado na Cell Stem Cell (Ref.23) mostrou que uma família de proteínas conhecidas como KZFP (Krüppel-associated box-containing zinc finger proteins) são fatores chaves na regulação e na domesticação dos TEs. Quando o genoma do embrião humano é ativado pouco tempo depois do óvulo ser fertilizado pelo espermatozoide, transposons estão entre as primeiras sequências a serem expressas. Nesse sentido, as KZFPs agem rapidamente minimizando o impacto transcricional durante os primeiros estágios de embriogênese, permitindo que TEs sejam usados mais tarde para o desenvolvimento e nos tecidos adultos.
EXEMPLO NOTÁVEL
Recentemente o genoma do tubarão-branco (Carcharodon carcharias) foi inteiramente sequenciado e descrito em uma publicação no periódico PNAS (Ref.36), revelando um tamanho genômico de 4,63 Gb (~1,5X o genoma humano), 24520 genes, 58,5% de sequências repetidas e a presença de uma enorme quantidade de transposons, especificamente do tipo LINE. É a mais alta proporção de LINEs (quase 30%) descoberta em vertebrados até o momento. Esse tipo de transposon - particularmente o LINE-1 - causa instabilidade genômica ao criar quebras de cadeia dupla no DNA, o que pode ter fomentado a evolução de vários mecanismos eficientes de reparação genética e de cura de feridas que são notáveis nesses predadores. No tubarão-branco, o LINE mais prevalente é o LINE-3/CR1, o qual parece possuir as mesmas capacidades de retro-transposição do LINE-1.
CONCLUSÃO
Existem vários mecanismos moleculares para a produção, deleção e acúmulo de novos genes, os quais baseiam e fomentam o processo evolutivo, e esses mecanismos frequentemente cooperam entre si para as transformações genômicas. Para o acúmulo de informações genéticas e consequente expansão genômica, a duplicação genética (mutação), a transposição de elementos e a transferência lateral de genes (compartilhamento genético) são os mais importantes desses mecanismos. Os padrões no material genético dos seres vivos não pode ser explicado fora de um contexto evolutivo.
REFERÊNCIAS CIENTÍFICAS
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5577496/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4236023/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4281893/
- https://www.ncbi.nlm.nih.gov/books/NBK21830/
- http://www.academicjournals.org/article/article1386259192_Roy%20and%20Deo.pdf
- https://www.ncbi.nlm.nih.gov/pubmed/26424194
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3420103/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6069113/
- https://www.ncbi.nlm.nih.gov/pubmed/20946257
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5590085/
- https://www.nature.com/articles/srep42571
- https://academic.oup.com/jhered/article-abstract/109/2/117/4417594
- https://www.ncbi.nlm.nih.gov/pubmed/28672159
- https://www.ncbi.nlm.nih.gov/pubmed/28672159
- https://www.ncbi.nlm.nih.gov/pubmed/30567990
- http://science.sciencemag.org/content/360/6393/eaar6343
- https://www.nature.com/articles/s41586-018-0734-6
- https://www.nature.com/articles/s41559-019-0822-5
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2874221/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4196381/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6240941/
- https://www.sciencedirect.com/science/article/pii/S1934590919301110
- https://www.cell.com/cell-stem-cell/pdf/S1934-5909(19)30111-0.pdf
- https://www.cell.com/cell/fulltext/S0092-8674(18)30843-2
- https://www.pnas.org/content/116/10/4400
- https://science.sciencemag.org/content/361/6403/eaar7191
- https://science.sciencemag.org/content/361/6403/eaar6089
- https://science.sciencemag.org/content/357/6346/93.abstract
- https://www.nature.com/articles/s41467-018-05051-5
- https://www.nature.com/articles/s41467-018-06108-1
- https://www.cell.com/iscience/fulltext/S2589-0042(18)30115-9
- https://elifesciences.org/articles/42153
- https://science.sciencemag.org/content/362/6412/343
- https://www.pnas.org/content/116/14/6908
- https://www.nature.com/articles/s41588-018-0153-5
- https://www.pnas.org/content/116/10/4446
- https://nph.onlinelibrary.wiley.com/doi/full/10.1111/nph.15715
- https://journals.sagepub.com/doi/full/10.1177/1559325819843376
- https://mbio.asm.org/content/10/3/e00837-19
- Martin et al. (2020). Genome ancestry mosaics reveal multiple and cryptic contributors to cultivated banana. The Plant Journal, Volume 102, Issue 5, Pages 1008-1025. https://doi.org/10.1111/tpj.14683
- Sardos et al. (2022). Hybridization, missing wild ancestors and the domestication of cultivated diploid bananas. Frontiers in Plant Science. https://doi.org/10.3389/fpls.2022.969220
- Slesak et al. (2011). Bowel obstruction from wild bananas: A neglected health problem in Laos. Tropical Doctor, 41: 85–90. https://doi.org/10.1258/td.2011.100293
- Roberts et al. (2021). Syncytins expressed in human placental trophoblast. Placenta, Volume 113, Pages 8-14. https://doi.org/10.1016/j.placenta.2021.01.006
- Meyer et al. (2017). Endogenous Retroviruses: With Us and against Us. Frontiers in Chemistry. https://doi.org/10.3389/fchem.2017.00023
- Frank et al. (2022). Evolution and antiviral activity of a human protein of retroviral origin. Science, Vol. 378, Issue 6618, pp. 422-428. https://doi.org/10.1126/science.abq7871
- Gophan & Altman-Price (2022). Horizontal Gene Transfer in Archaea—From Mechanisms to Genome Evolution. Annual Review of Microbiology, Vol.76:481-502. https://doi.org/10.1146/annurev-micro-040820-124627
- Borodovich et al. (2022). Phage-mediated horizontal gene transfer and its implications for the human gut microbiome, Gastroenterology Report, Volume 10, goac012. https://doi.org/10.1093/gastro/goac012
- Irwin et al. (2022). Systematic evaluation of horizontal gene transfer between eukaryotes and viruses. Nat Microbiol 7, 327–336. https://doi.org/10.1038/s41564-021-01026-3
- Melkonian et al. (2019). Genomes of Subaerial Zygnematophyceae Provide Insights into Land Plant Evolution. Cell, Volume 179, Issue 5, Pages 1057-1067.e14. https://doi.org/10.1016/j.cell.2019.10.019
- Huang et al. (2022). Major episodes of horizontal gene transfer drove the evolution of land plants. Molecular Plant, Volume 15, Issue 5, P857-871. https://doi.org/10.1016/j.molp.2022.02.001
- Dunning et al. (2021). Widespread lateral gene transfer among grasses. New Phytologist, Volume230, Issue6, Pages 2474-2486. https://doi.org/10.1111/nph.17328
- Hackl et al. (2023). Novel integrative elements and genomic plasticity in ocean ecosystems. Cell, Volume 186, Issue 1, P47-62.E16. https://doi.org/10.1016/j.cell.2022.12.006